picrust2功能预测-从qiime2安装到数据分析_picrust2功能预测结果怎么看-程序员宅基地
技术标签: github linux picrust2 windows
记录一下跌跌撞撞地摸索过程
picrust2功能预测
参照公众号《宏基因组》刘永鑫的原创:《PICRUSt2:OTU或ASV等16S随便预测宏基因组且数据库增加10倍》https://blog.csdn.net/woodcorpse/article/details/89302863
注:命令中 \为断行用,实际上输入命令要去掉,在输入文件路径时没有用此符号断行。
虚拟机与qiime2安装
参照公众号《宏基因组》刘永鑫的原创:《QIIME 2用户文档. 01简介和安装 Introduction & Install(2020.2) 》使用VirtualBox方式安装部分
https://blog.csdn.net/woodcorpse/article/details/75103929
下载最新版本虚拟机:https://download.virtualbox.org/virtualbox/6.1.4/VirtualBox-6.1.4-136177-Win.exe
下载最新版本虚拟机扩展包:
https://download.virtualbox.org/virtualbox/6.1.4/Oracle_VM_VirtualBox_Extension_Pack-6.1.4.vbox-extpack
下载QIIME2镜像:https://data.qiime2.org/distro/core/2020.2
安装:
1) 安装最新版虚拟机,默认位置,装好后将扩展包拷贝到安装目录windows/program files/oracle/virtualbox,双击安装扩展包。
2) 双击下载好的QIIME2镜像或在虚拟机界面点击导入镜像,修改分配给虚拟机的cpu和内存(一般是windows的一半),点击导入。
3) 启动虚拟机,菜单中安装Guest Additions,按照提示完成后,重启linux系统才能设置共享文件夹和共享复制粘贴。https://mp.weixin.qq.com/s/WS9u0nhiS1eizL5KXKs__A?
4) 设置共享文件夹,添加共享文件夹,勾选固定分配/自动挂载。
问题1:安装好后,输入qiime的命令均显示command not found。
前几天看到有人在百度上发起了同样问题。不是安装有问题,是qiime的命令和qiime2的命令不兼容,因此使用qiime2进行16分析的命令可参照https://www.cnblogs.com/afeiyuanda/p/11037287.html
Picrust2安装
https://github.com/picrust/picrust2/官网有给出两种安装方法,建议第一种安装,不会出错。
安装好后第二次使用picrust,也要输入激活命令:
conda activate picrust2
或者
conda activate picrust
如果想要使用qiime2命令,则要先退出picrust2,输入失活命令:
conda deactivate
问题2:v2.3.2-b版本优化了匹配度,测序序列进行后续分析时都会出现这样的问题,stopping - all 234 input sequences aligned poorly to reference sequence(--min_align option specified a minimum proportion of 0.8 aligning to reference sequences
先反向互补完,再做功能预测就可以了,seqtk seq -r otus.fa.format.fasta > otus_rc.fa
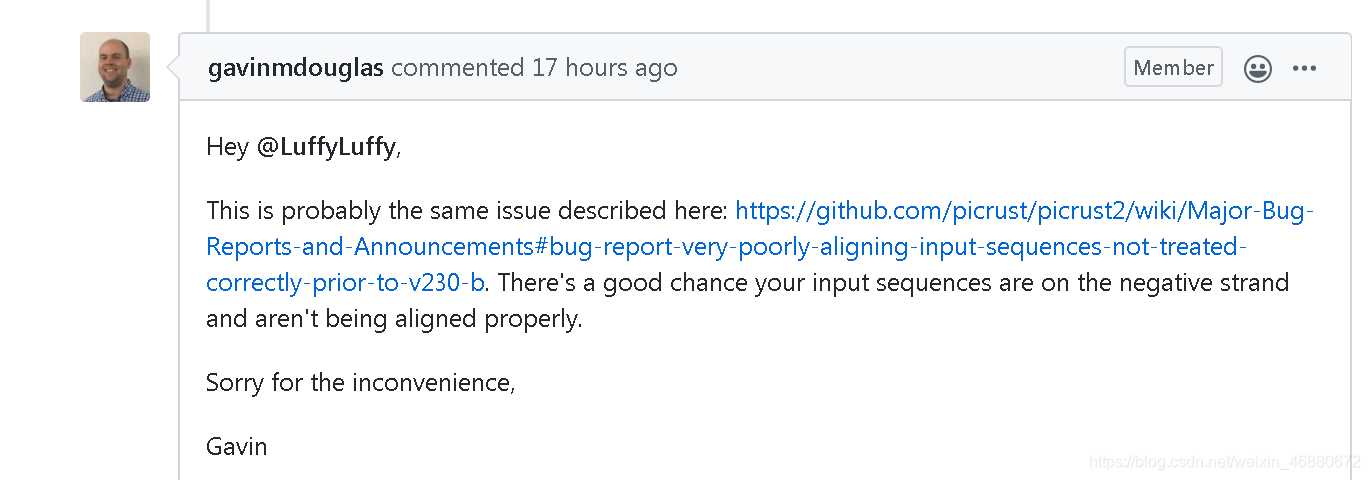
如果使用以前的版本不会出现这样的问题。
有个小插曲:不知道怎么解决这个问题的时候,无论输入什么数据都显示出这个结果,心态差点就崩了,然后花了点钱请淘宝上做生信分析的人,用我的数据帮我跑了一遍,可以出结果。最后观察版本之间有什么差别的时候就发现最新版本优化了,然后淘宝上那个生信技术厉害的客服用最新版本也出不了结果,他就去官网发了一个issue(就是上面图片里的),开发者很快就给回复了,解释了原因。
问题3:使用vsearch跑出的结果使用out.fa和out_table.biom两个文件可以直接跑出预测结果,但使用qiime跑出的结果中两个文件夹的out名称不一致,要删掉后面的其他字符可采用以下方法。
修改fastq文件OTU 名字`
awk '{print $1 }' FASTA_IN > FASTA_OUT
其中FASTA_IN和FASTA_OUT是输入和输出FASTA文件的名称。
$2:一行一行的读取指定的文件, 以空格作为分隔符,打印第二个字段
比如有这样一个文件
a1 b1 c1 d1
a2 b2 c2 d2
执行的结果是,输出:
b1
b2
问题4:会出现以下报错,大概率是电脑内存不够,picrust2官网有一个针对这个错误的issue。
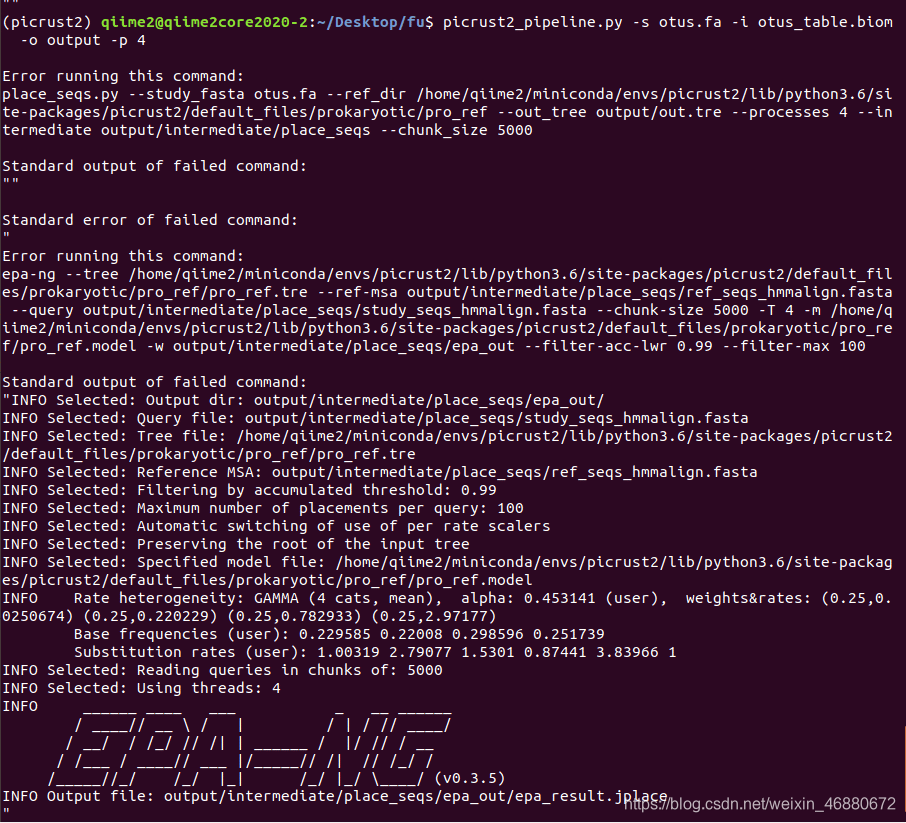
解决的根本办法是扩大你的内存,建议32G往上走,windows32G内存一般能够给虚拟机分配20G左右运行内存,在样本量比较多的情况下,往往16G是不够的,但是有时候样本量可以的情况下可以打擦边球试运行,降低CPU线程。我之前运行过分配给虚拟机36G的内存(windows总共48G)运行104个样本,使用4和2线程均无法得到结果,之后再使用1个线程耗时19000秒就成功了。也有试过88个样本,2线程9000秒。
###picrust2预测
比较完整的一步脚本是
picrust2_pipeline.py -s dna-sequences.fasta -i feature-table.biom -o \
picrust2_out_pipeline -p 20 -r default_files/prokaryotic/pro_ref/pro_ref \
--in_traits COG,EC,KO,PFAM,TIGRFAM
可能default_files/prokaryotic/pro_ref/pro_ref这个文件需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
这个命令运行后的结果包括COG,EC,KO,PFAM,TIGRFAM的丰度信息和MetaCyc途径丰度。
我一般输入的命令是:
picrust2_pipeline.py –s out.fa -i out_table.biom \
-o picrust2_out_pipeline -p 4
-p 4是使用4线程的意思
输出的结果包括EC,KO,PATHWAY的丰度信息。
但是但是结果文件中的结果比较简洁,比如只输入EC1.1.1,而不标注对应的酶名字,这时候需要加工加工。根据需要酌情添加描述:
add_descriptions.py -i EC_metagenome_out/pred_metagenome_unstrat.tsv \
-m EC -o EC_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i KO_metagenome_out/pred_metagenome_unstrat.tsv \
-m KO -o KO_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i COG_metagenome_out/pred_metagenome_unstrat.tsv \
-m COG -o COG_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i PFAM_metagenome_out/pred_metagenome_unstrat.tsv \
-m PFAM -o PFAM_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i TIGRFAM_metagenome_out/pred_metagenome_unstrat.tsv\
-m TIGRFAM -o TIGRFAM_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i pathways_out/path_abun_unstrat.tsv
-m METACYC -o pathways_out/path_abun_unstrat_descrip.tsv
结果输出中没有KEGG结果,可通过以下命令生成KEGG pathway 丰度表和注释结果
pathway_pipeline.py -i KO_metagenome_out/pred_metagenome_unstrat.tsv \
-o KEGG_pathways_out --no_regroup --map \
/home/qiime2/miniconda/envs/picrust/lib/python3.6/site-package/
picrust2/default_files/pathway_mapfiles/KEGG_pathways_to_KO.tsv
这里KEGG_pathways_to_KO.tsv需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
# 添加功能描述
add_descriptions.py -i KEGG_pathways_out/path_abun_unstrat.tsv.gz \
--custom_map_table /home/qiime2/miniconda/envs/picrust/lib/python3.6/site-
package/picrust2/default_files/description_mapfiles/KEGG_pathways_info.tsv.gz \
-o KEGG_pathways_out/path_abun_unstrat_descrip.tsv.gz
这里/KEGG_pathways_info.tsv.gz需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
如果不懂可看原文https://www.jianshu.com/p/e3beaee77423
或如果不想一步生成结果,就看官网分步命令:https://github.com/picrust/picrust2/wiki/PICRUSt2-Tutorial-(v2.1.4-beta)
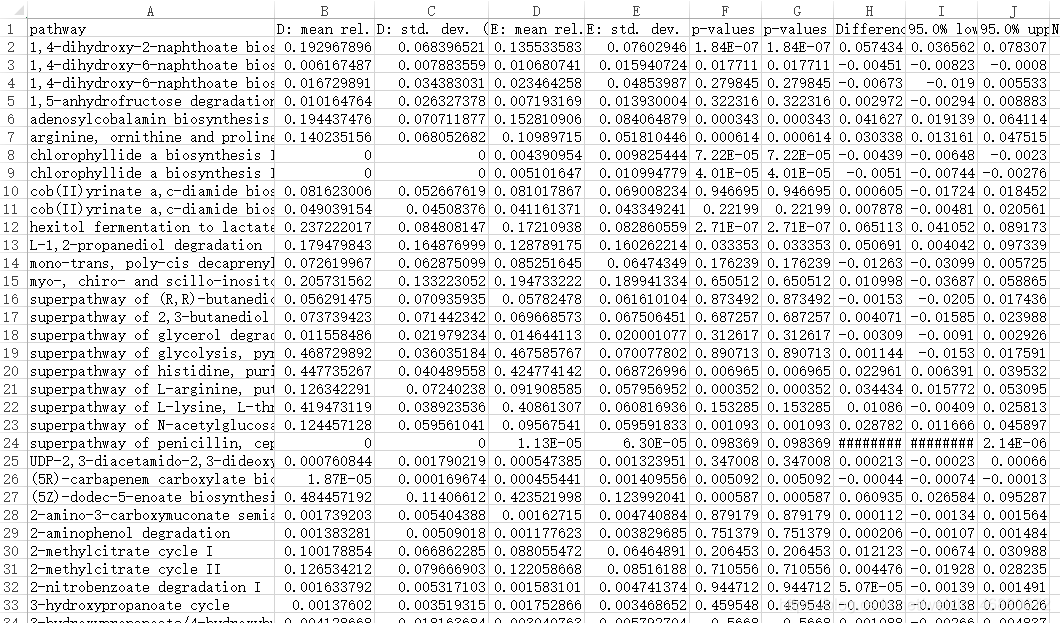
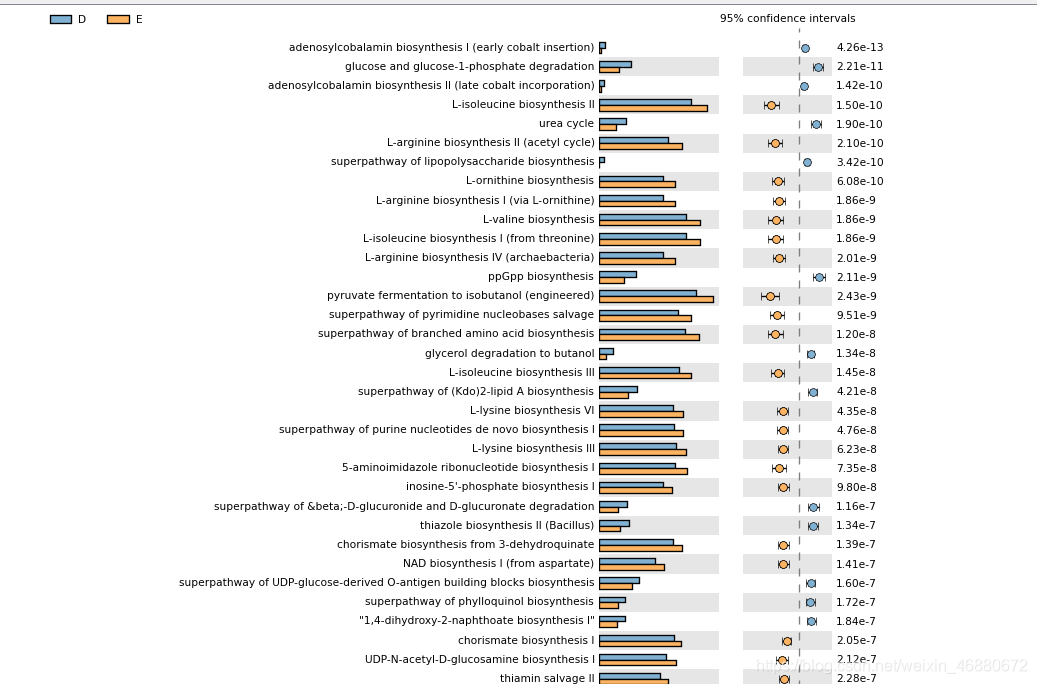
分析好的结果,使用STAMP软件进行显著性差异分析,该软件导入相应数据后既可以直接出很直观漂亮的显著性差异图,也可一次性导出所有通路或酶的组间差异分析详细数据。如下两图所示。
智能推荐
oracle 12c 集群安装后的检查_12c查看crs状态-程序员宅基地
文章浏览阅读1.6k次。安装配置gi、安装数据库软件、dbca建库见下:http://blog.csdn.net/kadwf123/article/details/784299611、检查集群节点及状态:[root@rac2 ~]# olsnodes -srac1 Activerac2 Activerac3 Activerac4 Active[root@rac2 ~]_12c查看crs状态
解决jupyter notebook无法找到虚拟环境的问题_jupyter没有pytorch环境-程序员宅基地
文章浏览阅读1.3w次,点赞45次,收藏99次。我个人用的是anaconda3的一个python集成环境,自带jupyter notebook,但在我打开jupyter notebook界面后,却找不到对应的虚拟环境,原来是jupyter notebook只是通用于下载anaconda时自带的环境,其他环境要想使用必须手动下载一些库:1.首先进入到自己创建的虚拟环境(pytorch是虚拟环境的名字)activate pytorch2.在该环境下下载这个库conda install ipykernelconda install nb__jupyter没有pytorch环境
国内安装scoop的保姆教程_scoop-cn-程序员宅基地
文章浏览阅读5.2k次,点赞19次,收藏28次。选择scoop纯属意外,也是无奈,因为电脑用户被锁了管理员权限,所有exe安装程序都无法安装,只可以用绿色软件,最后被我发现scoop,省去了到处下载XXX绿色版的烦恼,当然scoop里需要管理员权限的软件也跟我无缘了(譬如everything)。推荐添加dorado这个bucket镜像,里面很多中文软件,但是部分国外的软件下载地址在github,可能无法下载。以上两个是官方bucket的国内镜像,所有软件建议优先从这里下载。上面可以看到很多bucket以及软件数。如果官网登陆不了可以试一下以下方式。_scoop-cn
Element ui colorpicker在Vue中的使用_vue el-color-picker-程序员宅基地
文章浏览阅读4.5k次,点赞2次,收藏3次。首先要有一个color-picker组件 <el-color-picker v-model="headcolor"></el-color-picker>在data里面data() { return {headcolor: ’ #278add ’ //这里可以选择一个默认的颜色} }然后在你想要改变颜色的地方用v-bind绑定就好了,例如:这里的:sty..._vue el-color-picker
迅为iTOP-4412精英版之烧写内核移植后的镜像_exynos 4412 刷机-程序员宅基地
文章浏览阅读640次。基于芯片日益增长的问题,所以内核开发者们引入了新的方法,就是在内核中只保留函数,而数据则不包含,由用户(应用程序员)自己把数据按照规定的格式编写,并放在约定的地方,为了不占用过多的内存,还要求数据以根精简的方式编写。boot启动时,传参给内核,告诉内核设备树文件和kernel的位置,内核启动时根据地址去找到设备树文件,再利用专用的编译器去反编译dtb文件,将dtb还原成数据结构,以供驱动的函数去调用。firmware是三星的一个固件的设备信息,因为找不到固件,所以内核启动不成功。_exynos 4412 刷机
Linux系统配置jdk_linux配置jdk-程序员宅基地
文章浏览阅读2w次,点赞24次,收藏42次。Linux系统配置jdkLinux学习教程,Linux入门教程(超详细)_linux配置jdk
随便推点
matlab(4):特殊符号的输入_matlab微米怎么输入-程序员宅基地
文章浏览阅读3.3k次,点赞5次,收藏19次。xlabel('\delta');ylabel('AUC');具体符号的对照表参照下图:_matlab微米怎么输入
C语言程序设计-文件(打开与关闭、顺序、二进制读写)-程序员宅基地
文章浏览阅读119次。顺序读写指的是按照文件中数据的顺序进行读取或写入。对于文本文件,可以使用fgets、fputs、fscanf、fprintf等函数进行顺序读写。在C语言中,对文件的操作通常涉及文件的打开、读写以及关闭。文件的打开使用fopen函数,而关闭则使用fclose函数。在C语言中,可以使用fread和fwrite函数进行二进制读写。 Biaoge 于2024-03-09 23:51发布 阅读量:7 ️文章类型:【 C语言程序设计 】在C语言中,用于打开文件的函数是____,用于关闭文件的函数是____。
Touchdesigner自学笔记之三_touchdesigner怎么让一个模型跟着鼠标移动-程序员宅基地
文章浏览阅读3.4k次,点赞2次,收藏13次。跟随鼠标移动的粒子以grid(SOP)为partical(SOP)的资源模板,调整后连接【Geo组合+point spirit(MAT)】,在连接【feedback组合】适当调整。影响粒子动态的节点【metaball(SOP)+force(SOP)】添加mouse in(CHOP)鼠标位置到metaball的坐标,实现鼠标影响。..._touchdesigner怎么让一个模型跟着鼠标移动
【附源码】基于java的校园停车场管理系统的设计与实现61m0e9计算机毕设SSM_基于java技术的停车场管理系统实现与设计-程序员宅基地
文章浏览阅读178次。项目运行环境配置:Jdk1.8 + Tomcat7.0 + Mysql + HBuilderX(Webstorm也行)+ Eclispe(IntelliJ IDEA,Eclispe,MyEclispe,Sts都支持)。项目技术:Springboot + mybatis + Maven +mysql5.7或8.0+html+css+js等等组成,B/S模式 + Maven管理等等。环境需要1.运行环境:最好是java jdk 1.8,我们在这个平台上运行的。其他版本理论上也可以。_基于java技术的停车场管理系统实现与设计
Android系统播放器MediaPlayer源码分析_android多媒体播放源码分析 时序图-程序员宅基地
文章浏览阅读3.5k次。前言对于MediaPlayer播放器的源码分析内容相对来说比较多,会从Java-&amp;gt;Jni-&amp;gt;C/C++慢慢分析,后面会慢慢更新。另外,博客只作为自己学习记录的一种方式,对于其他的不过多的评论。MediaPlayerDemopublic class MainActivity extends AppCompatActivity implements SurfaceHolder.Cal..._android多媒体播放源码分析 时序图
java 数据结构与算法 ——快速排序法-程序员宅基地
文章浏览阅读2.4k次,点赞41次,收藏13次。java 数据结构与算法 ——快速排序法_快速排序法